Resumen 
La
investigación se realizó en base a la caracterización
morfológica de 235 entradas colectadas en el sur del país;
mismas que fueron analizadas con el paquete estadístico SAS
(Sistema de Análisis Estadístico), utilizando el
agrupamiento jerárquico de Ward sobre la base de datos
morfológicos, se obtuvieron grupos que corresponden a cada
especie taxonómicamente identificada. La caracterización
molecular se realizó utilizando la técnica RAPD
(Polimorfismos de ADN amplificados al Azar) que consta de tres pasos:
la extracción de ADN, se logró gracias a la combinación
de los protocolos de Ferreira - Grattapaglia (1998) y, Del Río
y Bambey (1993); la cuantificación se realizó
utilizando el protocolo del DENAREF, comparando la intensidad de sus
bandas con las del estándar ADN Low Mass Ladder (10068-013,
GIBCO BRL); para la amplificación se realizó un sondeo
preliminar probando 98 primers de Operon Technologies, en cinco
entradas de especies diferentes; de éstos
se seleccionaron 21 que amplificaron productos polimórficos y
reproducibles, mismos que fueron evaluados en las 29 entradas; se
obtuvo como resultado 205 bandas, las más
intensas y polimórficas que presentaron amplificación.
El análisis de agrupamiento molecular se realizó a
partir de la matriz de similitud, usando el método de
agrupamiento UPGMA, con el coeficiente Jaccard del paquete
estadístico NTSYSpc, que sirve para determinar la similitud
genética entre accesiones; los dendrogramas resultantes
muestran tres ramas principales que agrupan especies de acuerdo a su
clasificación taxonómica, éstos son similares al
fenograma construido en base a datos morfológicos pero que
varían en su topología. Se realizó también
un análisis de coordenadas principales (PCO) en base a datos
moleculares, que evidencia las relaciones genéticas entre
especies. Las correlaciones entre caracteres morfológicos y
moleculares resultaron altamente significativos y, corroboran la
coherencia entre estos y su eficacia para caracterizar germoplasma.
El análisis filogenético se hizo sobre la base binaria
de datos morfológicos y moleculares, utilizando el programa
PAUP (Sistema de Análisis Estadístico) para determinar
las relaciones de parentesco inter e intraespecífica; el
cladograma obtenido se apoya en un grupo externo (C. papaya),
evidenciando así las relaciones genéticas entre
especies. Como resultado de la investigación se evidencia las
relaciones inter e intra específicas y, la gran variabilidad
de genotipos dentro del género en estudio, siendo la provincia
de Loja la más diversa. La condición de alogamia, así
como la facilidad de cruzamiento entre especies, y las constantes
mutaciones somáticas en la propagación vegetativa,
aseguran el aparecimiento de nuevos genotipos que amplían la
base genética del género Vasconcellea.
Palabras clave:
caracterización morfológica, genética,
sur-Ecuador
Introducción 
La familia Caricaceae con seis géneros, encierra diversidad de especies y subespecies; siendo individuos de polinización cruzada dan origen a nuevos genotipos; estas especies constituyen un potencial alimenticio y económico muy importante, debido al contenido de vitaminas, proteínas y elementos indispensables para el organismo, propiedades organolépticas deseadas, así como, fuente de material promisorio para fitomejoradores. Desafortunadamente aún falta mucho por investigar ya que la mayoría de especies se encuentran en estado silvestre y amenazadas por un alto grado de erosión genética; la papaya (C. papaya), en la actualidad es la única entidad taxonómica del género Carica debido a la categorización reciente del grupo Vasconcellea; el género Vasconcellea actualmente comprende 21 especies, por ende es el mas representativo de la familia, tienen su origen en Sudamérica a diferencia de la papaya que según reportes se originó en Centroamérica (Kim et al. 2002); entre las especies mas conocidas y comercializadas tenemos: el babaco (Vasconcellea x heilbornii var. pentagona) y en menor grado papayuela (V. pubescens) conocida como papaya chilena que constituyen una entrada económica considerable para el productor.
Hasta el momento pocos estudios a nivel molecular han sido realizados, el primer trabajo para el estudio de las relaciones genéticas entre especies de Carica y Vasconcellea fue emprendido por Jobin-Decor et al. (1997) utilizando marcadores isoenzimáticos y moleculares (RAPDs), el mismo que evidencia la divergencia genética entre estos dos tipos de especies; Van Droogenbroeck et al. (2002) pone en evidencia un proceso de introgresión bidireccional como factor responsable de la amplia variabilidad de ciertos genotipos híbridos de Vasconcellea. Bajo este precepto, la investigación se realizó con el fin de determinar: La distribución geográfica, la variabilidad y la relación de parentesco entre especies de Vasconcellea; para ello se realizó una caracterización morfológica y molecular de individuos colectados en la región sur del país; además se colectó germoplasma de reproducción sexual y asexual, para establecer una colección de campo. Estas actividades se desarrollan con el fin de conservar un componente de la biodiversidad y poseer un conocimiento más amplio de las especies, que sirvan de base para posteriores trabajos de mejoramiento, así como para plantear alternativas que conduzcan a proteger estos recursos genéticos.
Materiales y Métodos 
Colecta y caracterización
La colecta se realizó en las provincias de: Loja, Zamora Chinchipe, El Oro y parte del Azuay. Para la caracterización morfológica, en cada sitio de caracterización se escogieron al azar cinco plantas por especie y se aplicó la guía de descripción que incluye 150 variables. La base de datos para el análisis estadístico se construyó a partir de 119 variables morfológicas que aportaron mayor información: 66 cuantitativas y 53 cualitativas. Utilizando el paquete SAS versión 6.12 (SAS Institute Inc 1990) y la distancia de Gower (1967) se estimó la similitud taxonómica entre cada par de entradas para caracteres continuos, mientras que para los caracteres cualitativos se utilizó el coeficiente de asociación entre entradas. La estructura taxonómica de las entradas fue analizada por medio del agrupamiento jerárquico de Ward (1963), que hace posible encontrar en cada estado aquellos dos grupos cuya unión produzca el mínimo incremento en la suma total de cuadrados del error dentro de grupos. La elección del número de grupos de entradas se realizó con los criterios de Pseudo F y Pseudo t2, utilizando el procedimiento CLUSTER del paquete estadístico SAS, mediante este análisis se reconoció dentro del grupo de caracteres utilizados, aquellos con mayor valor discriminante y que, por lo tanto permitieron una eficiente identificación de la relación entre los clones o entradas de la población en estudio, para un determinado carácter y para el grupo de caracteres. El valor discriminante para separar grupos se estimó sobre la base del análisis de frecuencias y las estadísticas de Cramer (Kendall & Stuart 1979), coeficiente de asociación (Fienberg 1977) y Chi cuadrado (X2; Cochran 1954).
La distribución geográfica y los sitios de mayor variabilidad de las entradas del género Vasconcellea del sur del Ecuador, se determinaron utilizando el programa DIVA versión 2; se ingresaron datos de nueve variables multiestados: forma de la hoja, color por dentro y por fuera de flor masculina y femenina, color de la piel del fruto, forma transversal y longitudinal del fruto, color de la pulpa y, los datos de longitud y latitud (transformados a unidades de distancia), para cada una de las 235 entradas.
Para la caracterización molecular se seleccionaron 29 entradas de las 235, dos entradas de cada especie y algunos ecotipos de V. heilbornii, los más diferentes morfológicamente. Esta caracterización requiere de los siguientes pasos: 1) Para la extracción de ADN, se probaron algunos protocolos, hasta que finalmente con la combinación de los protocolos de Ferreira - Grattapaglia (1998) y, Del Río y Bambey (1993), se llegó a definir el protocolo de extracción de ADN para las especies en estudio, el mismo que se detalla a continuación.
- Verter 0,5 g de tejido foliar deshidratado y macerado, en tubos eppendorf.
- Adicionar antioxidantes: B-mercaptoetanol y bisulfito de sodio en dosis de 100 ul y 0,2 g respectivamente, agitar hasta que la mezcla se torne homogénea; posteriormente agregar 1 ml de solución tampón de extracción (CTAB 2X) y agitar nuevamente.
- Incubar los tubos durante 2 horas a 650C en baño maría agitando cada 10 minutos para homogeneizar la suspensión.
- Retirar los tubos, dejar enfriar a temperatura ambiente y, centrifugar a 14000 rpm durante 20 min.
- Transferir el sobrenadante a un tubo nuevo y adicionar 600 ul de CIA (Cloroformo : Alcohol Isoamílico 24:1), agitar suavemente y centrifugar a 14 000 rpm durante 5 min.
- Capturar la fase superior sin perturbar la interfase y depositar a un tubo nuevo.
- Adicionar luego a la fase acuosa 1 ml de solución tampón de precipitación (2X), mezclar suavemente e incubar a temperatura ambiente por 30 min, luego centrifugar a velocidad máxima durante 5 min y eliminar el sobrenadante.
- A la pastilla de ADN que se encuentra en el fondo del tubo adicionar 400 l de solución Tampón TE (Tris HCl EDTA), una vez resuspendido el pellet se colocar 200 l de acetato de amonio 7,5 M y 400 l de etanol 95%; mezclar suavemente y dejar precipitar los ácidos nucleicos durante 20 min, centrifugar a velocidad máxima y eliminar el sobrenadante y dejar secar el ADN durante la noche.
- Finalmente resuspender la pastilla con 50 - 100 l de solución tampón (TE 0.1 M).
- Si el ADN se muestra contaminado, aplicar la técnica de purificación (Jhigan 1992).
2) Cuantificación, la integridad y concentración del ADN fueron analizadas por electroforesis en minigeles de agarosa y cuantificadas comparativamente por la intensidad de sus bandas con las del estándar ADN Low Mass Ladder (10068-013, GIBCO BRL) utilizando el protocolo del laboratorio del DENAREF. Para ello se preparó un gel de agarosa 0,8 % (0,25 g de agarosa, 25 ml de buffer TAE 1X y 1,25 ul de Etbr 1X), una vez listo el gel se mezclaron en parafilm: 4 l de cada ADN (muestra), con 1 l de loading (blue juice manufacturado SIGMA). El volumen de carga fue 2 l a partir del segundo poso, una vez cargadas todas las muestras, se colocó en el primer poso 2 l de marcador ADN Low Mass Ladder (10068-013, GIBCO BRL). La electroforesis se realizó a 80 V por 30 min. Seguidamente se llevó el gel a la cámara de luz ultravioleta en un fotodocumentador (UVP Gel Documentation System), focalizando bien la imagen (intensidad de luz y tiempo de exposición) para luego imprimirla en papel térmico (SIZE 50 es suficiente para la cuantificación). Una vez obtenido ADN de buena calidad se procedió a degradar el ARN, adicionando 1 l de RNAse (R-4642,SIGMA) en 100 l de solución (10 l/ml). La concentración de ADN de trabajo de cada una de las muestras se estandarizó diluyéndolo en tampón TE 0,1 M tartrazine, hasta lograr una concentración final de 5 ng/l.
3) Para la amplificación, se realizó un sondeo preliminar utilizando el protocolo de la Universidad de Wisconsia modificado por Williams, probando 98 primers, para ello se escogieron cinco muestras, de las especies más diferentes morfológicamente; como resultado de este proceso se seleccionaron 21 primers que presentaron polimorfismos reproducibles y confiables. En tubos PCR colocar una alícuota de 3 l de ADN muestra (5 ng/l). También preparar una mezcla con los siguientes componentes: 5X buffer (66,00 l), Primer (1,0 uM) (12,00 l), DNTPs (2,5 mM cada uno) (12,00 ul), Taq polimerasa (5 U/ul) (3,90 ul) y, agua ultra pura (69,00 ul). Posteriormente extraer 5,43 l de la mezcla y colocar en cada uno de los 29 tubos PCR, la reacción resultante cubrirla con una gota de aceite mineral, para evitar la evaporación de los elementos. Estas reacciones fueron amplificadas en un termociclador MJ Research PTC - 100, de acuerdo al siguiente programa: ciclo inicial de desnaturalización a 94 oC por 5 min, 40 ciclos de desnaturalización cíclica a 94oC por 1 min, 1 min de anillamiento a 36oC, 2 min de elongación cíclica a 72oC, y un ciclo final de elongación a 72oC por 7 min. usando el 1kb DNA Ladder Marker (15615-016, GIBCO BRL) como marcador de referencia, los productos de amplificación se analizaron por electroforesis en geles de agarosa al 2%, en tampón TAE 1X con bromuro de etidio (10 mg/ml, GIBCO BRL) a una concentración final de 500 mg/l en el gel. La electroforesis se realizó a 110 V durante 3 horas y, finalmente las bandas de ADN fueron visualizadas bajo luz ultravioleta en un fotodocumentador (UVP Gel Documentation System), e impresas en papel térmico. La codificación de cada banda polimórfica se representa por medio de los estados presencia (1) o ausencia (0), también se tomaron en cuenta polimorfismos que involucraban datos dudosos (9), los cuales se registraron por inspección en una matriz de datos, en el programa Ntedit del paquete estadístico NTSYS, pc ver. 2,0 (Applied Biostatistics Inc, 1998). Los tamaños de las bandas polimórficas se calcularon mediante la aplicación Length (Templeton 1988) y se expresaron en pares de bases (pb). La naturaleza binaria de los datos exigió la utilización de un coeficiente de asociación para cuantificar la similitud entre dos muestras.
Sobre la matriz obtenida se aplicó un análisis de agrupamiento o Cluster Análisis, empleando el método de agrupamiento UPGMA cuyo objeto es sintetizar la información de todas las muestras; mediante la opción SAHN CLUSTERING del paquete NTSYSpc, con la opción TREE DISPLAY fueron visualizados los agrupamientos o relaciones entre las accesiones de la colección, generándose diagramas arborescentes o dendrogramas que mostraron las relaciones genéticas entre accesiones. El PCO (Gower 1966) es un método de ordenación homólogo al análisis de componentes principales (PCA) pero especialmente adaptado a datos cualitativos.
El análisis filogenético se realizó, utilizando la base de datos binaria morfológica y molecular en el programa PAUP (Análisis Filogenético Usando Parsimonia), versión 3.1.1. La opción ACCTRAN (Transformación Acelerada) fue escogida como un método de optimización del orden de los caracteres. La búsqueda heurística fue conducida usando la opción branch swapping, TBR (Árbol de bisección y reconstrucción) que permitió observar todos los posibles árboles parsimoniosos, para finalmente visualizar el cladograma; el análisis fue validado por el uso de un grupo externo (GE), C. papaya.
Resultados y Discusión 
Caracterizacion molecular
La colecta consta de 235 entradas provenientes de las provincias de Loja, Zamora Chinchipe, parte alta de El Oro y parte del Azuay. La mayoría de accesiones se registraron en la provincia de Loja. La colecta incluye accesiones de un rango altitudinal que va desde los 990 m, con la entrada 207 V. parviflora en la provincia de El Oro, Cantón y parroquia Zaruma, hasta los 2724 m, con la entrada 168, 169 y 170 V. heilbornii spp, en la provincia de Loja, parroquia Chuquiribamba
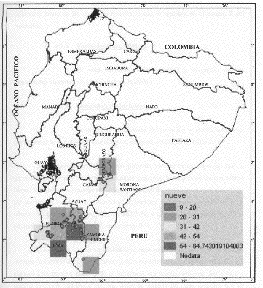
Figura 1.
Distribución geográfica de la colección de
especies del género Vasconcellea del Sur del Ecuador y
centros de mayor diversidad en base a caracteres multiestados. Los
códigos 9 a 64 indican el números de estados (Forma de
hojas, color de flores, forma y color de frutos).
Figure 1. Geographic distribution of Vasconcellea in Southern Ecuador. (-64 represent the number of stages (leaf form, flower color, from and color of fruits).
Figure 1. Geographic distribution of Vasconcellea in Southern Ecuador. (-64 represent the number of stages (leaf form, flower color, from and color of fruits).
La (Figura 1) muestra los sitios de mayor variabilidad de especies de Vasconcellea en el sur del Ecuador; la mayor diversidad está representada por el color rojo que agrupa de 54 a 64 estados de caracteres, seguido del color naranja con 42 a 54, luego el amarillo de 31 a 42, a continuación el verde claro con 20 a 31 y, finalmente el verde oscuro con 9 a 20 estados de caracteres. De acuerdo a estos resultados, la provincia de Loja es la más rica en diversidad de especies, pues esta zona goza de variedad de climas y microclimas, que se constituyen en el ecosistema ideal para el desarrollo de variedad de especies, subespecies y ecotipos de plantas.
Caracterizacion molecular
Extracción y cuantificación de ADN, con el protocolo de extracción se logró obtener ADN de buena calidad, sin degradación que migró en el gel, como banda única de alto peso molecular. Con la cuantificación realizada comparativamente con el estándar ADN Low Mass Ladder se obtuvieron concentraciones desde 20 a 50 ng/ul, con rendimientos de 2,0 a 5,0 ug.
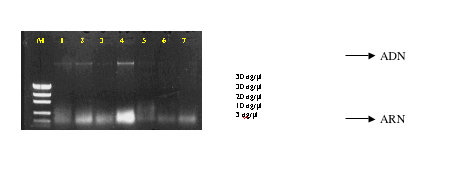
Figura
2. Cuantificación de ADN genómico total de especies del
género Vasconcellea. M: marcador DNA Low Mass Ladder
(GIBCO) cuyas concentraciones para cada banda se detallan a la
izquierda. Carriles 1-7 : DNA extraído de las muestras.
Figure 2. Quantification of total genome DNA in species of Vascioncellea. M: Low Mass Ladder. Row 1-7: Extracted DNA.
Figure 2. Quantification of total genome DNA in species of Vascioncellea. M: Low Mass Ladder. Row 1-7: Extracted DNA.
Productos de amplificación
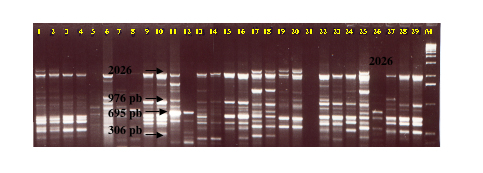
Figura
3. Perfiles RAPDs obtenidos en la amplificación en
Vaconcellea con el primer OPAC-19. Carriles 1 - 29: DNA total
amplificado. M = marcador. A la izquierda el tamaño en pares
de bases (pb) de algunas bandas polimórficas evaluadas.
Figure 3. RAPD profile after amplification with primer OPAC 19. Rows 1-29 show total amplified DNA. M = marker. On the left some polymorph bands.
Figure 3. RAPD profile after amplification with primer OPAC 19. Rows 1-29 show total amplified DNA. M = marker. On the left some polymorph bands.
De la amplificación de los 21 primers en las 29 entradas estudiadas, se registraron 205 bandaspolimórficas, que representan un 70,4% del total de bandas obtenidas, sin tomar en cuenta las borrosas o dudosas, sino únicamente las bandas intensas y polimórficas, las mismas que permitieron generar la matriz de datos para el análisis. El rango de los tamaños de amplificación por primer varió de 259 a 2455 pares de bases (pb) con una media de 10,4 polimorfismos/primer, de un total de 12 a 18 fragmentos amplificados/primer. Por otro lado, el rango del número de fragmentos RAPD amplificados varió entre dos y 18, con valores mínimo y máximo de 132 y 305 respectivamente.
Los valores de similitud obtenidos de la aplicación del coeficiente de Jaccard en las comparaciones pareadas de las 12 especies estudiadas variaron entre 0,12 y 0,44 con un promedio global de 0,39. Cabe indicar que este valor promedio fue el más bajo en el estudio conducido por Van Droogenbroeck et al. (2002) entre ocho taxa del género Vasconcellea, en el que el valor promedio global obtenido fue de 0,54. Esta diferencia, si bien no es extrema, puede explicarse por la cantidad de información molecular analizada en estos estudios, que desde esta perspectiva si es significativa (205 bandas RAPD generadas en el presente estudio vs. 496 fragmentos AFLP obtenidos por los laboratorios de la Universidad de Ghent).
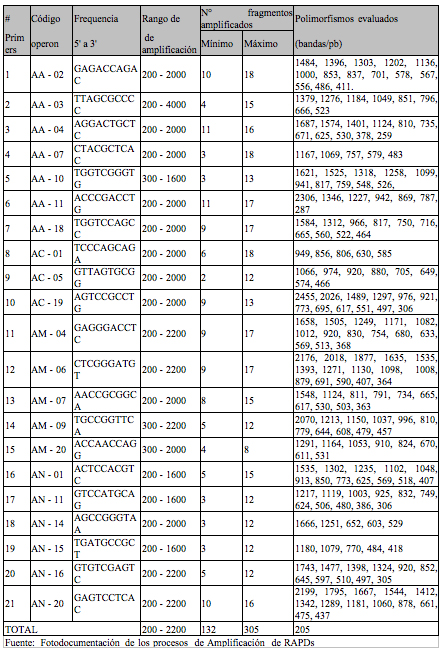
Tabla 1. Primers
(21) y productos polimórficos de amplificación
registrados en 27 accesiones Vasconcellea y 2 de Carica
del Sur de Ecuador.
Table 1. Primers (21) and polymorph products of registered amplification in 27 accessions of Vasconcellea and 2 of Carica in Southern Ecuador.
Table 1. Primers (21) and polymorph products of registered amplification in 27 accessions of Vasconcellea and 2 of Carica in Southern Ecuador.
Similitud Genética
En cuanto a la variabilidad intraespecífica, los valores de similitud obtenidos (en cursiva), muestran a V. heilbornii como la especie de mayor heterogeneidad genética al presentar el valor más bajo (0,59); este valor puede deberse a dos factores: a) un mayor número de muestras de esta especie fueron incluidas en el estudio (n = 8) y, b) la naturaleza genética de los materiales (origen híbridos o introgresiones). El resto de especies presentó altos valores de similitud (>90), lo que demuestra una mayor uniformidad genética intraespecífica si bien el número de muestras no se puede considerar altamente representativo. Sin embargo, especies como V. palandensis y C papaya mostraron valores más (90 y mas) sugiriendo un grado estimable de variabilidad genética. En cuanto al grado de similitud interespecífica, el valor más alto resulta de la comparación de V. pubescens y la especie no identificada taxonómicamente V. sp (0,62), mientras que entre V. pubescens y C. papaya se obtuvo el valor más bajo (0,12), lo cual refleja el grado de heterogeneidad genética existente ente estos dos taxa.
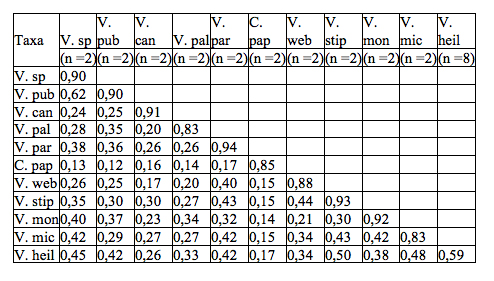
Tabla
2. Valores de similitud genética obtenidos mediante el
coeficiente de Jaccard entre y dentro de las 12 especies incluidas en
el presente estudio.
Table 2. Value of genetic similarity by Jaccard coefficient in and betweeb 12 species included in this study.
Table 2. Value of genetic similarity by Jaccard coefficient in and betweeb 12 species included in this study.
Análisis de Agrupamiento (Cluster
analysis)
Los valores de similitud obtenidos en comparación dos a dos entre las entradas estudiadas varían entre 0,16 y 0,94; con un promedio de 0,55.
Método UPGMA
El dendrograma generado a partir del análisis UPGMA dividió a las 28 entradas en tres ramas o grupos principales, representados con los códigos A, B, C. La primera rama (A) está formada por 13 accesiones que corresponden a especies colectadas en Loja y Azuay. La rama B incluye 13 entradas provenientes de Loja, Zamora, El Oro y Azuay. La rama última rama (C) incluye dos entradas colectadas en Loja.
En el árbol UPGMA se distinguen tres ramas principales, en donde C. papaya (rama C) por encontrarse en la parte basal del dendrograma, es la especie más distante del conjunto; esto contrasta con estudios realizados con marcadores RFLPs, RAPDs e izoenzimas que produjeron 137 fragmentos polimórficos entre C. papaya y el resto de especies (Vasconcellea), y produjeron un índice de disimilitud del 70%; como resultado de ello C. papaya formó un grupo monofilético. Actualmente el genero Carica lo forma la especie C. papaya que tuvo su origen en Centro América; el resto de especies corresponden al género Vasconcellea originarias de Sudamérica (Alvarez 2002).
Por otro lado, la rama B la constituyen en su mayoría especies silvestres (excepto V. monoica y V. heilbornii 204); el análisis mostró una fuerte asociación entre V. monoica (187 y 190), con V. heilbornii (204) y probablemente entre éstas especies pudo haber ocurrido introgresión. Así por ejemplo, molecularmente comparten bandas de ADN, mientras que morfológicamente las características en común son: color blanquecino de la flor, haz brillante de la hoja y fruto consistente, entre otras.
En la rama A se encuentran especies cultivadas, de frutos comestibles: V. stipulata, V. pubescens V. heilbornii, que pueden ser el resultado de la selección del hombre; además se encuentra la especie silvestre no identificada taxonómicamente (1 y 2); ésta última especie es polígama, de pedúnculos largos, frutos pequeños con escasa pulpa y abundante semilla, genéticamente cercana a V. pubescens aunque no comparten en lo absoluto similitud a nivel morfológico.
En esta rama las accesiones de V. heilbornii forman subgrupos, reflejando posiblemente casos de introgresión en su genoma; puesto que son especies alógamas (dioicas, monoicas y polígamas), de fácil cruzabilidad, han generado híbridos con grados variables de fertilidad; según Alvarez (2002), estos híbridos ocurren espontáneamente en áreas donde la distribución de la especie es abundante. Los híbridos de V. heilbornii se encuentran generalmente junto a sus progenitores, estas plantas parecen ser el resultado del cruzamiento natural entre V. pubescens y V. stipulata.
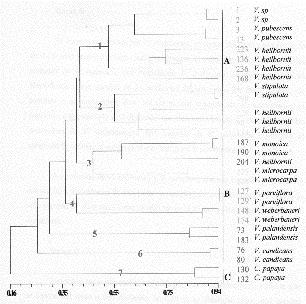
Figura 4.
Dendrograma en base a datos moleculares de entradas del género
Vasconcellea usando el coeficiente de Jaccard y el método
de agrupamiento UPGMA. Las letras A, B y C representan cada una de
las ramas principales del árbol. Los códigos 1 a 7
representan el número de subgrupos.
Figure 4. Dendrogram based on molecular data from the genus Vasconcellea using Jassard's coefficient and UPGMA grouping. Letters A, B and C represent each main branch of the tree. Number 11-7 represent the subgroups.
Figure 4. Dendrogram based on molecular data from the genus Vasconcellea using Jassard's coefficient and UPGMA grouping. Letters A, B and C represent each main branch of the tree. Number 11-7 represent the subgroups.
En el árbol UPGMA, se observa que las especies cultivadas tienden a agruparse entre sí, lo cual evidencia la similitud genética entre éstas. Las entradas de V. candicans (76 y 80) por encontrarse en la base de las ramas A y B, se consideran las más primitivas en la historia evolutiva de las Vasconcelleas en estudio; morfológicamente esta especie difiere del resto por contener plantas arbóreas, de hojas enteras acorazonadas, con ramas laterales múltiples y, frutos de piel rugosa. Hay que anotar que el árbol NJ tiene similar estructura que el árbol anterior. Por otro lado se distinguen tres ramas principales, en donde la rama C, es igual a la del árbol UPGMA; la rama B, formada por V. candicans y V. palandensis que son las especies ancestrales de las Vasconcelleas en estudio; la rama A, agrupa el resto de especies silvestres y cultivadas, en donde V. pubescens y V. stipulata posibles progenitores de V. heilbornii se encuentran separados por especies silvestres, lo que indica que posiblemente los híbridos V. heilbornii, de fácil cruzabilidad, han intercambiado material genético con otras especies de Vasconcelleas. Así mismo este dendrograma (NJ) muestran a V. heilbornii distribuida en tres subgrupos, reflejando posiblemente casos de introgresión en su genoma.
Es evidente que los marcadores moleculares ayudaron a complementar la interpretación de las relaciones genéticas entre especies de los géneros Carica y Vasconcellea y, a agrupar las entradas de acuerdo a su similitud. Los estudios demostraron las variaciones RAPDs presentes entre y dentro de las accesiones estudiadas, permitiendo así examinar la diversidad genética y las relaciones filogenéticas entre taxa. Los análisis Neighbour-Joining y UPGMA de una matriz de similitud de fragmentos RAPDs, produjeron fenogramas similares en su topología, agrupando cada entrada de acuerdo a la clasificación taxonómica, por tanto contienen grupos de taxa similares; sin embargo algunas diferencias importantes fueron evidentes en los dos fenogramas.
Análisis de Coordenadas Principales
(PCO)
En la figura 5 se muestra el PCO en función de los dos primeros vectores "eigen", aquí se representa la dispersión de las entradas de los géneros Carica y Vasconcellea, la cual permite determinar las relaciones genéticas existentes entre especies. La variabilidad representada por los dos primeros vectores "eigen" es alta y el valor observado (25,3 %) es superior al valor estadístico esperado (24,4 %), por lo que estos vectores fueron utilizados con validez para analizar la distribución genética del germoplasma en estudio. La técnica PCO permitió definir la diversidad genética y observar una alta similitud jerárquica con los dendrogramas NJ y UPGMA. El vector 1 ubica a C. papaya como la especie más distante del estudio, lo que concuerda con la separación taxonómica de esta especie y, el resto de especies como Vasconcelleas en otra unidad taxonómica. Dentro del grupo de Vasconcelleas se muestra a V. candicans como la más distante del conjunto.
Análisis fitogenetico
El PCO apoya los resultados obtenidos en el Cluster analysis y, ayuda a diferenciar claramente los géneros Carica y Vasconcellea. Las entradas de Vasconcellea presentan estrecha similitud genética, además esta técnica ilustra las relaciones genéticas entre grupos; así como también la relación genética entre V. stipulata, V. pubescens y los híbridos descendientes; pues muestra claramente que las accesiones del híbrido natural: V. heilbornii se ubican entre los posibles padres: V. stipulata, y V. pubescens. La accesión de V. heilbornii que comparte material con V. monoica, refleja la diversidad existente en el grupo de accesiones, y por ser individuos de polinización cruzada es posible asumir que ha existido recombinación genética entre especies (introgresión natural).

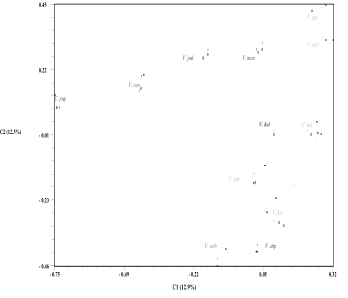
Figura
5. Distribución en el plano definido por los dos primeros
vectores "eigen" del PCO basado en el coeficiente de
similitud de Jaccard en las comparaciones pareadas de dos entradas de
Carica y 26 de Vasconcelleas con 205 polimorfismos
RAPDs. El porcentaje de la varianza expresada se indica bajo el eje
correspondiente. El código 1, 2, 3 y 4 corresponden a los
grupos definidos por el "cluster análisis".
Figure 5. Eigenvalue distribution of PCO based on Jassard's similarity coefficient comparing the entries of Carica and 26 Vasconcella samples with 205 RAPD polymorphisms. The percentage of variance is inicated on the respective axis. 1-4 corresponds to the groups defined by cluster analysis.
Figure 5. Eigenvalue distribution of PCO based on Jassard's similarity coefficient comparing the entries of Carica and 26 Vasconcella samples with 205 RAPD polymorphisms. The percentage of variance is inicated on the respective axis. 1-4 corresponds to the groups defined by cluster analysis.
El cladograma resultante del análisis filogenético posee una longitud de 1527 pasos, formado por cuatro (1, 2, 3 y 4) clados, y un grupo externo (C. papaya); se obtuvo un índice de retención (RI) de 0,586 que representa un valor medio de similaridad de los taxa y en particular de las sinapomorfias de los cuatro clados. Adicionalmente el cladograma presenta un índice de homoplasia (HI) de 0,713 y, de consistencia (CI) 0,287 que son relativamente bajos y permiten formular hipótesis sobre la validez de los datos RAPD's para filogenia.
El cladograma dividió los taxa en cuatro clados, con una raíz que la constituyó C. papaya (GE). Estos cuatro clados son: el clado I que agrupa taxas provenientes de diferentes sitios de las provincias de Loja, Azuay y El Oro; el clado II está formado por especies colectadas en Loja, El Oro, Zamora Chinchipe y Azuay; además el clado III agrupa dos accesiones endémicas de Zamora Chinchipe; y el último clado (IV), está formado por dos accesiones colectadas en diferentes sitios de la provincia de Loja. papaya
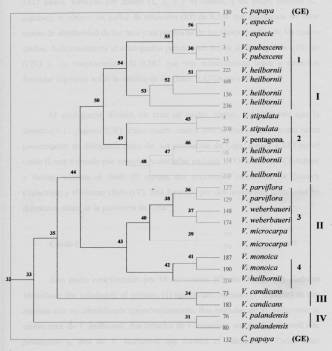
Figura 6.
Cladograma de tres árboles parsimoniosos generados de la
matriz de datos del análisis RAPDs y de datos morfológicos
de 27 entradas de Vasconcellea, usando como grupo externo a C.
papaya. Los números en las ramas indican el número
de pasos evolutivos. Los códigos I, II, III y IV son el número
de ramas asignadas.
Figure 6. Cladogram of tree parsimoneous trees generated from the data matriz of the RAPD análisis of 27 samples of Vasconcellea, using C. papaya as extreme group. The branch numbers indicate the number of evolutionary steps. Coses I-IV are branch numbers.
Figure 6. Cladogram of tree parsimoneous trees generated from the data matriz of the RAPD análisis of 27 samples of Vasconcellea, using C. papaya as extreme group. The branch numbers indicate the number of evolutionary steps. Coses I-IV are branch numbers.
El grupo externo está formado por dos entradas de C. papaya, que produjeron una dicotomía en la base del árbol. Sin embargo, los dos materiales no resuelven el mismo clado, lo cual refleja cierto grado de variabilidad intraespecífica en C. papaya.
Cuando se utiliza como grupo externo a C. papaya, obtenemos que la especie más primitiva es V. candicans, luego sigue V. palandensis, (cuyas diferencias morfológicas están en sus foliolos), siendo enteras acorazonadas para el primer caso y digitadas para el segundo; luego se encuentra un clado formado en su mayoría por especies silvestres; finalmente en la rama más evolucionada se encuentran especies cultivadas, además de accesiones de V. heilbornii que forma varios subgrupos que se ubican junto a sus posibles progenitores: V. stipulata y V. pubescens.
Utilizando como grupo externo a C. papaya, (aunque el estudio incluye solo dos genotipos de V. candicans), El cladograma mostró que V. candicans es la especie más distante, por lo que en la historia evolutiva del género Vasconcellea, V. candicans habría sido la primera especie en desarrollarse; igual ilustración se muestra en el dendrograma utilizando el método de agrupamiento UPGMA, así como en el PCO; esto se corrobora con los estudios realizados por Van Droogenbroeck et al. (2002) que menciona que de acuerdo a las observaciones morfológicas, es la única especie arbórea, que forma hojas enteras y racimos laterales múltiples.
Así mismo, esta figura ilustra que entre las especies ancestrales del conjunto de Vasconcellea en estudio, V. candicans, se caracteriza por tener hojas enteras y ramas laterales múltiples; y V. palandensis, que presenta hojas digitadas. Luego se encuentra el grupo de especies silvestres: V. microcarpa, V. weberbaueri, V. parviflora, que son plantas de frutos pequeños, con abundante semilla y escasa pulpa, no útiles para el consumo humano; además en este clado se encuentra un subgrupo (4) formado por V. monoica y, una entrada de V. heilbornii, que al parecer comparten material genético, reflejando así la diversidad genética entre grupos.
En este mismo cladograma las entradas de C. papaya no resuelven el mismo clado, lo cual refleja cierto grado de variabilidad intraespecífica dentro del género Carica. Se ilustra además un alto nivel de diversidad intraespecífica en V. heilbornii, que incluye a V. heilbornii pentagona e híbridos no identificados taxonómicamente; esta diversidad también fue observada por Aradhya et al. (1999), quienes señalan que por la condición de alogamia, pudo haber ocurrido más de una cruza recíproca entre los padres V. stipulata y V. pubescens. Otras explicaciones incluyen la incorporación de plantas originadas de semillas producidas por recombinación sexual desconocida, o por acumulación de mutaciones somáticas a través de la propagación vegetativa.
Alvarez (2002) menciona que en el caso de V. pubescens, por la variación sexual descrita (monoecia y dioecia), unida a la capacidad de formar híbridos con otras especies, otorga la posibilidad de generar nuevas combinaciones e incrementar la variabilidad; muchos parientes silvestres de la papaya son intercompatibles y se pueden cruzar y polinizar para producir híbridos con grados variantes de fertilidad; estos híbridos ocurren espontáneamente en áreas donde la distribución de la especie es abundante. Todo esto contribuye a que cada vez aparezcan nuevos genotipos que amplían la base genética del género Vasconcellea.
Hay que tomar en cuenta, que el método de caracterización molecular RAPD se basa en el estudio del ADN genómico total, el cual está en constante proceso de recombinación genética, por tanto no es el más adecuado para un estudio filogenético, más bien debe visualizarse como una herramienta de apoyo para el mismo; además, la gran diversidad del género Vasconcellea, así como la condición de alogamia, que permiten el cruzamiento de las especies, dan origen a nuevos genotipos que amplían la base genética de este género. Se supone que de esta gran diversidad de genotipos, algunos posiblemente ya se han extinguido y otros, que no han sido considerados en el análisis, contribuyen con la baja confiabilidad de los cladogramas.
En definitiva este trabajo complementa y corrobora en esencia los resultados recientemente publicados por Van Droogenbroeck et al. (2002) indicando que existe una gran variabilidad de genotipos y una estructuración genética consistente dentro del género Vasconcellea, así como la divergencia de este con la papaya (C. papaya).
Referencias 
Alvarez, L. 2002. Colección, Caracterización y Evaluación de papayas de altura, con énfasis en papayuelas. Departamento de Recursos Naturales, Facultad de Ciencias Agropecuarias, Universidad de Caldas, Colombia.
Badillo, V. 1971. Monografía de la Familia Caricaceae. Universidad Central de Venezuela. Facultad de Agronomía. Maracay, Venezuela. p. 152 -159.
Badillo, V. 1999. Familia Caricaceae. 2 ed. Universidad Central de Venezuela. Facultad de Agronomía. Maracay, Venezuela. p. 36 -101.
Crisci, J. 1983. Introducción a la teoría y práctica de la taxonomía numérica. Washington D.C. p. 36-67.
Del Rio, C.A. 1990. Análisis de la variación isoenzimática de Oxalis tuberosa Molina "Oca" y su distribución geográfica. Universidad Ricardo Palma. Tesis Lic. Biol. 61 p.
Estrella, J. 1998. Molecular Systematic of the Neotropical Tuberosus Legumet Pachyrhizus. Rich. Ex DC. The Yam Beam. School of Environmental and Evolutionary Biology. A thesis submitted to the University of ST. Andrews for the Degree of Doctor of Philosophy. St. Andrews. p. 100-200.
Ferreira, M.Y. & D. Grattapaglia, D. 1998. Introducción al uso de marcadores moleculares en el análisis genético. EMBRAPA-CENARGEN, Brasilia, Brasil. p. 38-56
Gower, J. 1967. A comparison of some methods of cluster analysis. Biometrics, 23:623-637.
INIAP. 2000. Protocolos de laboratorio de marcadores moleculares copilados por Morillo E. biólogo responsable del laboratorio de la EESC. DENAREF.
Jhingan, A. 1992. A novel technology for DNA isolation. Methods Mol. Cell. Biol., 3: 15-22
Kendall, M. & A. Stuart. 1979. The advanced theory of statistics. Volumen 2 New York: Macmillan Publishing Company, Inc.
Piedra, A. 2002. Caracterización Morfológica y Molecular de la colección Nacional de Oca Oxalis tuberosa del banco de germoplasma del INIAP. Tesis de Grado. Quito, Ecuador. p. 22 - 70.
SAS INSTITUTE. 1990. SAS/STAT user's guide, 6th ed. SAS Institute, Cary, NC.
Van, B; P. Droogenbroeck & P. Breyne 2002. AFPL analysis of genetic relationships among papaya and its wild relatives (Caricaceae) from Ecuador.
Ward, J. 1963. Hierarchical grouping to optimize an objective function. Journal of the American Statistical Association, 58:236-244.